近日,我校药学院、生物反应器工程国家重点实验室、光遗传学与合成生物学交叉学科研究中心杨弋教授、朱麟勇教授、浙江大学生命科学研究院任艾明研究员课题组联合在荧光RNA研究中取得突破性进展,在国际权威学术期刊《自然—化学生物学》(Nature Chemical Biology)在线发表题为“Structural basis of a small monomeric Clivia fluorogenic RNA with large Stokes shift” 的研究文章。
RNA具有独特的三维结构和种类繁多的生物学功能,广泛参与细胞的各类生命活动,与多种疾病的发生与发展密切相关。荧光RNA是近些年来发展起来的活细胞RNA成像技术。与荧光蛋白一样,研究人员只需要将荧光RNA的编码序列与靶标RNA编码序列融合,在细胞内表达后,即可对活细胞RNA进行标记成像。2023年杨弋、朱麟勇、陈显军以及任艾明课题组联合攻关,创新发展了Clivia系列大斯托克斯位移荧光RNA,具有高稳定性、高信噪比、高亮度,是目前唯一可用于活细胞分析的大斯托克斯位移荧光RNA,也是唯一可在活体上对RNA动态进行测量的荧光RNA(Nature Methods, 2023, 20, 1563)。然而,Clivia和荧光染料NBSI的识别和发色机制仍不清晰,这个谜底的揭示不仅对于Clivia荧光RNA的进一步优化和应用拓展具有重要意义,还可以为更多具有特殊性能荧光RNA的创造提供新的思路。
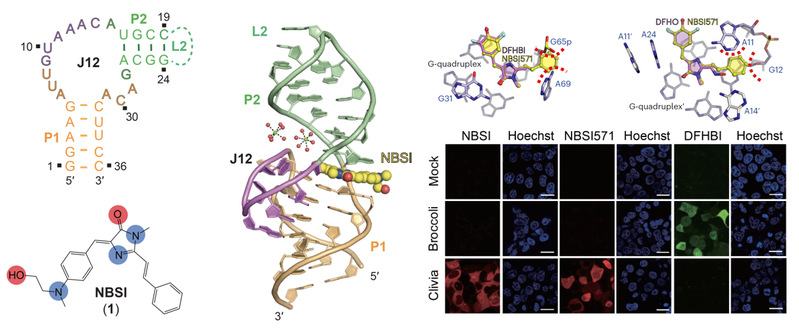
针对上述关键科学问题,研究团队对Clivia-NBSI复合物的三维结构进行了解析。结果显示,Clivia-NBSI整体结构非常紧凑,呈纺锤型,含有两个双螺旋茎区P1(橘色)和P2(绿色),以及一个的连接P1 和P2内环J12(紫色)和一个发卡环L2(绿色)。J12中A11、A12和A13与双螺旋P1通过远距离相互作用形成三个连续堆积的三碱基平面,不但稳定了整体RNA分子结构,还为配体结合口袋的形成提供了重要的结构支持。配体结合口袋位于三碱基平面上方,在整体结构的中间位置,P1的三碱基平面U8-A29-C30 和P2的碱基对G28-A15将染料配体NBSI像“三明治”一样夹在两层平面之间,J12的G9和C14从侧面将染料配体NBSI进一步固定在结合口袋内。配体与结合口袋之间通过完美的形状互补实现特异性识别,参与识别配体的关键碱基均在突变荧光实验中得以验证。该结构中鉴定的多层三碱基平面为进一步理解和探索荧光RNA适配体潜在的通用结构提供了重要线索。此外,作为RNA分子结构,复合物结构中镁离子和水分子也参与了维持整体结构的稳定以及配体结合口袋的形成。基于Clivia独特的配体识别模式及对染料NBSI化学结构的分析,通过结构的系统比较与分子对接模拟,该研究还验证了Clivia-NBSI系列荧光RNA可以与其他荧光RNA如Spinach-DFHBI,Broccoli-DFHBI以及Corn-DFHO等正交使用,实现多种RNA分子的同时标记与成像。
论文第一作者为黄凯怡、宋倩倩和方梦悦博士,通讯作者为杨弋教授、朱麟勇教授和任艾明研究员。该研究得到了国家重点研发计划、国家自然科学基金等经费资助。
原文链接:https://www.nature.com/articles/s41589-021-00884-6。